Ativação do proteassomo 20S aumenta tempo de vida cronológico de leveduras
Em células eucarióticas, o proteassomo é o complexo proteico responsável pela degradação de proteínas danificadas e não funcionais, regulando diversos processos celulares, como metabolismo, sinalização, divisão e morte celular. O acúmulo de proteínas danificadas, dentre as quais as oxidadas, está relacionado ao envelhecimento e a doenças neurodegenerativas. Estudos têm mostrado que a integridade do proteassomo é necessária para que as células se recuperem de situações de estresse ou tenham o tempo de vida aumentado.
Em um estudo realizado com leveduras Saccharomyces cerevisiae mutantes, o grupo da pesquisadora Marilene Demasi, do Instituto Butantan e do CEPID Redoxoma, mostrou que a ativação do proteassomo 20S via abertura da câmara catalítica está relacionada com o aumento do tempo de vida cronológico da levedura e maior resistência ao estresse oxidativo. Foram estudadas linhagens de S. cerevisiae com mutações nos resíduos de cisteína e de serina na subunidade alfa-5 do proteassomo 20S.
Segundo Demasi, este trabalho revela um sofisticado mecanismo regulador que controla a abertura do proteassomo 20S e afeta o tempo de vida da levedura, baseado nas interações entre vários aminoácidos da subunidade alfa-5. “Já havíamos identificado as cisteínas da subunidade alfa-5 que são modificadas por um processo redox chamado glutatiolação e, desta forma, promovem a abertura do proteassomo 20S. Agora, fizemos leveduras mutantes para tornar inequívoca a importância das modificação redox dessas cisteínas para a sobrevivência das células”.
O artigo, publicado na revista Archives of Biochemistry and Biophysics, contou a colaboração dos grupos dos pesquisadores Mario H. Barros, do ICB-USP, e Luis E.S. Netto, do IB-USP, ambos do CEPID Redoxoma, Daniel C. Pimenta, do Instituto Butantan, e Cristiano L.P. Oliveira, do IF-USP. O CEPID Redoxoma é um Centro de Pesquisa, Inovação e Difusão financiado pela FAPESP.
Tempo de vida cronológico
A degradação de proteínas ocorre por um processo sinalizado pela proteína ubiquitina. As proteínas oxidadas, no entanto, são degradadas de maneira independente de ubiquitinação, pelo proteassomo 20S livre — a unidade catalítica do proteassomo não acoplada a unidades regulatórias. O proteassomo 20S tem uma estrutura na forma de barril, com quatro anéis heptaméricos: dois anéis beta internos, que formam o centro catalítico onde ficam os sítios ativos, e dois anéis alfa nas extremidades, responsáveis pela abertura e fechamento da protease. Unidades reguladoras são acopladas a um ou a ambos os lados da estrutura central 20S, sendo a unidade 19S a mais abundante. Quando acoplado à unidade reguladora 19S, o proteassomo é chamado 26S. Estima-se que o proteassomo 20S livre represente mais de um terço do pool total de proteassomos das células de mamíferos e de leveduras. Além disso, já foi demonstrado que apenas 20% do pool contêm complexos com dois lados acoplados (19S-20S-19S), indicando que, na maioria desses complexos, a câmara catalítica 20S está livre.
Em 2012, o grupo de Demasi revelou pela primeira vez um mecanismo redox de regulação do proteassomo, mostrando que a S-glutationilação de dois resíduos de cisteína, Cys76 e Cys221, na subunidade alfa-5 promove a abertura do proteassomo 20S livre e, em consequência, o aumento da degradação de proteínas oxidadas.
Para esse novo estudo, os pesquisadores construíram linhagens de leveduras com mutações sítio-especificas nestas cisteínas anteriormente identificadas, que foram substituídas por serinas — α5-C76S e α5-C221S. “Fizemos mutação simples, em que apenas uma das cisteínas é substituída, e dupla mutação, substituindo ambas as cisteínas. O primeiro resultado que obtivemos foi que as células sem as duas cisteínas são inviáveis, o que demonstra a importância desses resíduos para o funcionamento adequado do proteassomo”, explicou Demasi. Além disso, as leveduras com mutações simples apresentaram menor tempo de vida cronológico e o proteassomo 20S predominantemente na conformação fechada.
O resultado mais interessante, no entanto, surgiu a partir de uma mutação randômica. Durante os experimentos realizados para obter a levedura com mutação da cisteína 221, os pesquisadores observaram que uma linhagem apresentava tempo de vida cronológico aumentado. Ao sequenciar o gene mutado, descobriram uma mutação secundária inesperada: a serina 35 havia sido substituída por uma prolina (S35P), juntamente com a esperada mutação C221S. A linhagem com a dupla mutação α5-S35P/C221S, com a cisteína 76 preservada, apresentou vida cronológica mais longa e maior resistência ao estresse oxidativo.
Para investigar esse resultado, os pesquisadores realizaram estudos fenotípicos e de estrutura do proteassomo. Utilizando a técnica de microscopia eletrônica de transmissão, observaram que na levedura dupla mutante a frequência com a qual o proteassomo é encontrado aberto é de 80%, enquanto na levedura selvagem a frequência é de 65%. No ensaio de envelhecimento cronológico (CLS, Chronological lifespan) a linhagem α5-S35P/C221S apresentou tempo de vida aumentado em 25%, em comparação com a linhagem selvagem, enquanto as linhagens α5-C76S e α5-C221S tiveram esse tempo diminuído em 50% e 25%, respectivamente. De acordo com a pesquisadora, esses resultados indicam que o aumento do tempo de vida da levedura se deve à abertura do proteassomo. Da mesma forma, quando as leveduras são tratadas com peróxido de hidrogênio, a linhagem duplo mutante é mais resistente ao estresse oxidativo por ter uma maior frequência do proteassomo na conformação aberta e, portanto, maior eficiência na remoção de proteínas oxidadas.
WT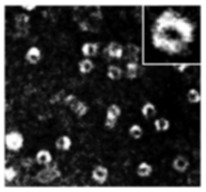 | α5-C76S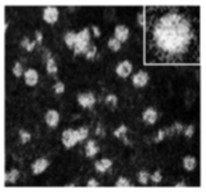 | α5-S35P/C221S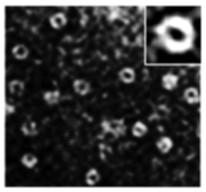 |
Microscopia eletrônica de transmissão mostra frequência do proteassomo 20S na conformação aberta em linhagens de levedura selvagem, com mutação na Cys 76 e com a dupla mutação S35P/C221S Adapted from Leme at al., 2019 (doi: 10.1016/j.abb.2019.03.012) | ||
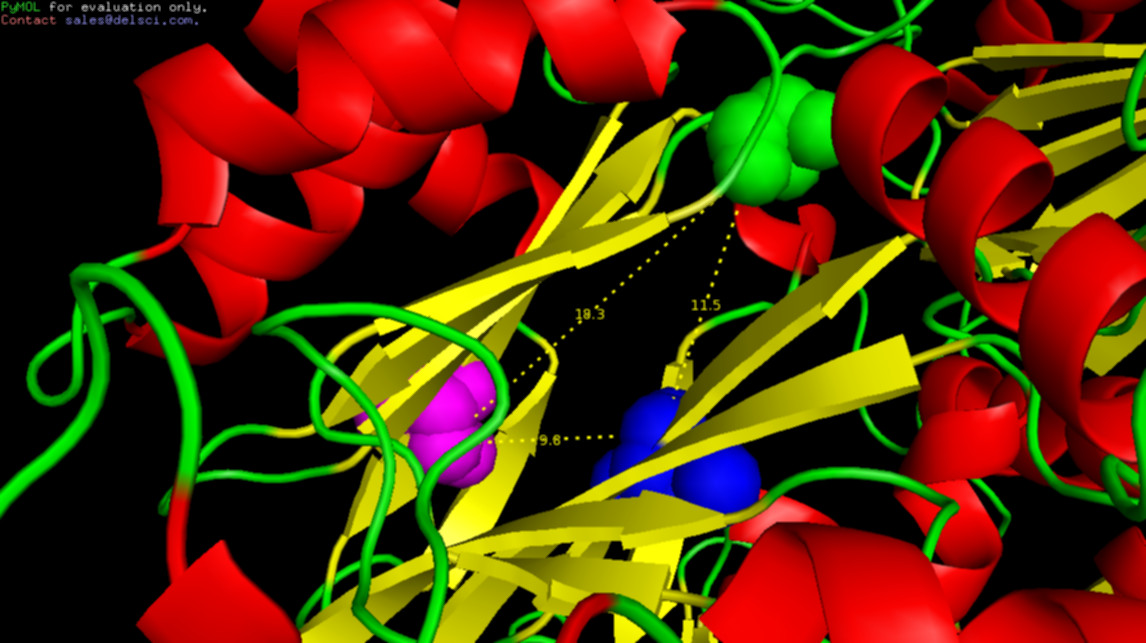
Demasi explica que, no proteassomo 20S com dupla mutação, a cisteína 76 está glutatiolada, como foi mostrado por espectrometria de massas. Quando esse proteassomo é tratado com um redutor, ou seja, quanto a glutationa é retirada, a frequência com a qual o proteassomo aparece aberto cai para 20%. No caso das linhagens com mutações simples, quando uma das cisteínas está mutada, a outra não sofre glutatiolização, o que indica que ambas são necessárias para que ocorra essa modificação. A Cys221 está localizada na superfície externa do proteassomo, sendo acessível ao solvente e, portanto, mais suscetível a uma modificação redox; a Cys76 está voltada para a parte central da subunidade, não sendo exposta ao solvente.
A hipótese dos pesquisadores é que quando a Cys221 é oxidada, ocorre uma mudança estrutural no proteassomo, que permite que a Cys76 também seja glutatiolada. No caso da dupla mutante α5-S35P/C221S, a prolina provavelmente induz uma mudança conformacional, expondo a Cys76 e, com isso, facilita sua glutatiolação e a consequente abertura do proteassomo. Análises por espectrometria de massas das mutantes mostraram que a Cys76 é o principal determinante para a abertura da câmera catalítica do proteossomo 20S e para sobrevivência celular Esta cisteína, lembra a pesquisadora, é altamente conservada ao longo da cadeia evolutiva, ocorrendo desde a levedura até o homem.
Segundo Demasi, os próximos passos da pesquisa envolvem estudos de dinâmica molecular e maior detalhamento da estrutura do proteassomo 20S por crio-microscopia eletrônica.
O artigo Mutations of Cys And Ser Residues in the Α5-Subunit of The 20s Proteasome from Saccharomyces Cerevisiae Affects Gating and Chronological Lifespan, de Janaína M. M. Leme, Erina Ohara, Verônica F. Santiago, Mario H. Barros, Luis E. S. Netto, Daniel C. Pimenta, Douglas O. C. Mariano, Cristiano L. P. Oliveira, Renata N. Bicev, Maria L. M. B. de Chaves, Caroline A. Lino e Marilene Demasi, pode ser acessado aqui.
